文献解读|Adv Sci(14.3):综合乳酸组学表征揭示胃肠道癌症中蛋白质调控的分子动力学
✦ +
+
论文ID
原名:Integrated Lactylome Characterization Reveals the Molecular Dynamics of Protein Regulation in Gastrointestinal Cancers
译名:综合乳酸组学表征揭示胃肠道癌症中蛋白质调控的分子动力学
期刊:Advance Science
影响因子:14.3
发表时间:2024.07.17
DOI号:10.1002/advs.202400227
背 景
胃肠道 (GI) 癌症是一组异质性恶性肿瘤,占癌症发病率的 26% 以上,占所有癌症相关死亡率的 35%。最近针对人类胃肠道癌症体的蛋白质基因组学图谱的研究揭示了肿瘤进展的广泛分子机制。然而,针对胃肠道癌症的靶向治疗方法有限,这凸显了开发用于诊断和治疗这种疾病的有效分子靶点的重要性。
代谢重编程是癌症的一个标志,肿瘤细胞主要依靠糖酵解来提供能量,并依靠代谢中间体来支持肿瘤的生长。已鉴定出以中间代谢物为底物的各种酰化修饰。组蛋白中乳酸衍生的赖氨酸乳酸化 (Kla) 已确定为一种新型的翻译后修饰 (PTM)。鉴于癌细胞中糖酵解增强和乳酸过量产生,乳酸化的发现为乳酸在癌症进展中的重要机制提供了新的视角。
实验设计
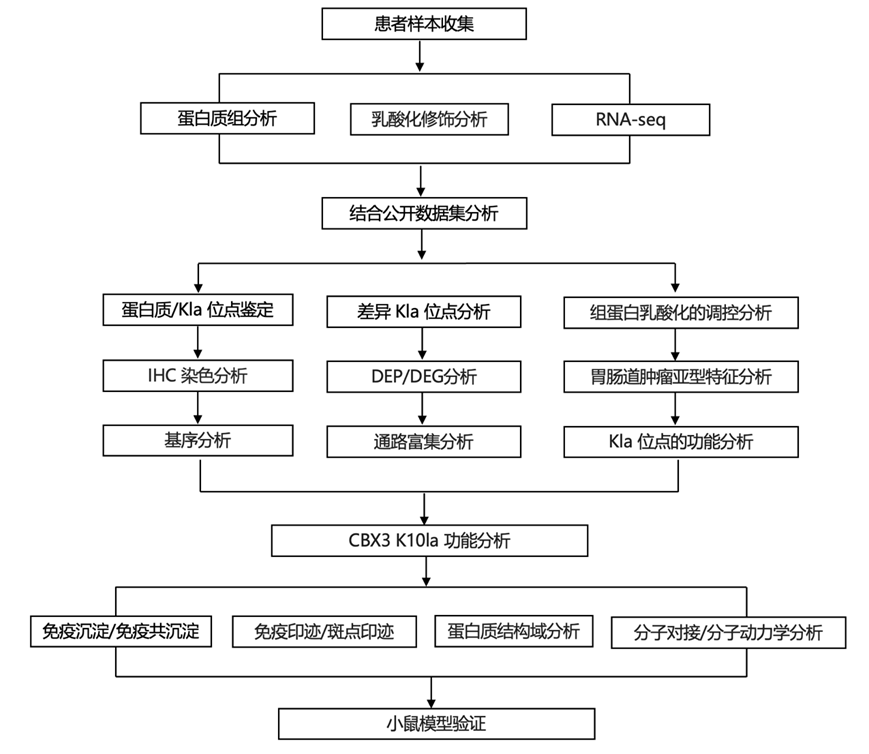
结 果
01
胃肠道癌症蛋白质组数据中广泛存在的赖氨酸乳酸化修饰
为了全面了解 Kla 在胃肠道肿瘤进展中的分子作用,研究团队收集了 40 例肝癌、胰腺癌、结直肠癌或胃癌患者的肿瘤和正常邻近组织 (NAT) 进行乳酸组和蛋白质组学分析(图 1A)。他们从 40 例 GI 肿瘤和 NAT 样本中分别鉴定了 9690 种蛋白质以及 3156 种蛋白质,这一共包含了 11698 个 Kla 位点,其中 6260 个 Kla 位点位于肝脏、3763 个位于胰腺、8100 个位于结直肠、5526 个位于胃组织。在肿瘤中鉴定的 Kla 位点和蛋白质的数量明显高于在配对非肿瘤组织中鉴定的 Kla 位点和蛋白质的数量(图1B-C)。与 NAT 组织相比,GI 癌症中乳酸化修饰的丰度和类型整体增加,并且侵袭性肿瘤具有更多的 Kla 修饰(图 1-E)。用泛乳酸化抗体对不同的 GI 肿瘤和 NAT 组织进行免疫组织化学 (IHC) 染色,结果显示癌症组织中的信号强度明显更高(图 1F),这表明 GI 蛋白质组在癌症组织中广泛发生了 Kla 修饰。
为了研究人类胃肠道组织中乳酸化修饰的氨基酸序列差异,他们对所有 11 616 个乳酸化肽段进行了 Kla 基序分析。使用热图评估 21 个氨基酸序列环境中 Kla 位点周围每个残基相对于人类胃肠道蛋白质组的差异性(图 1G)。丙氨酸和赖氨酸残基出现在乳酸化肽段的上游和下游,并且出现概率最高。半胱氨酸、亮氨酸和色氨酸残基在乳酸化肽段中出现的概率最低。五个最保守的基序是丙氨酸、赖氨酸、缬氨酸和甘氨酸,它们围绕着受体乳酸化赖氨酸,对乳酸化赖氨酸位点的分析表明,这些位点对丙氨酸具有强烈的偏好(图 1H)。
他们收集了本研究检测到的非组蛋白上的 Kla 位点,在本研究检测到的 11698 个 Kla 位点中,有 11571 个位点位于非组蛋白上,表明 Kla 是一种除组蛋白之外的普遍存在的修饰(图 1I)。乳酸化蛋白的亚细胞定位分析揭示了广泛的细胞影响,尤其是在细胞核和细胞质中,暗示 Kla 通过修饰细胞核和细胞质蛋白质发挥重要的生物学功能(图 1J)。接下来,他们进行蛋白质结构域和通路富集分析以分析Kla 的潜在功能,结果显示功能保守结构域和生物过程显著富集,包括基因调控、代谢、基因组稳定性和其他标志性癌症通路(图 1K-L)。
为了寻找全胃肠道组织中的 Kla 修饰模式,他们分析了从不同胃肠道样本中鉴定出的乳酸化位点之间的重叠情况,发现 1852 个 Kla 位点存在于所有四个队列中。值得注意的是,赖氨酸乳酸化修饰在每个胃肠道样本中也表现出特定的模式,其中仅在肝脏中鉴定出 2073 个赖氨酸乳酸化修饰,胰腺中鉴定出 499 个赖氨酸乳酸化修饰,结直肠中鉴定出 3195 个赖氨酸乳酸化修饰,胃样本中鉴定出 546 个赖氨酸乳酸化修饰(图 1M)。使用 DAVID 数据库进行深入的GO分析表明,这些在不同胃肠道癌症中常见或特定的乳酸化蛋白在不同的功能通路中富集,可能反映了它们在组织水平上特定调控中的作用(图 1N)。总的来说,这些据表明,广泛的 Kla 修饰涵盖了在四种类型的胃肠道癌症中具有广泛功能分布的蛋白质,这意味着 Kla 在胃肠道癌症发病机制中具有潜在的调控作用。
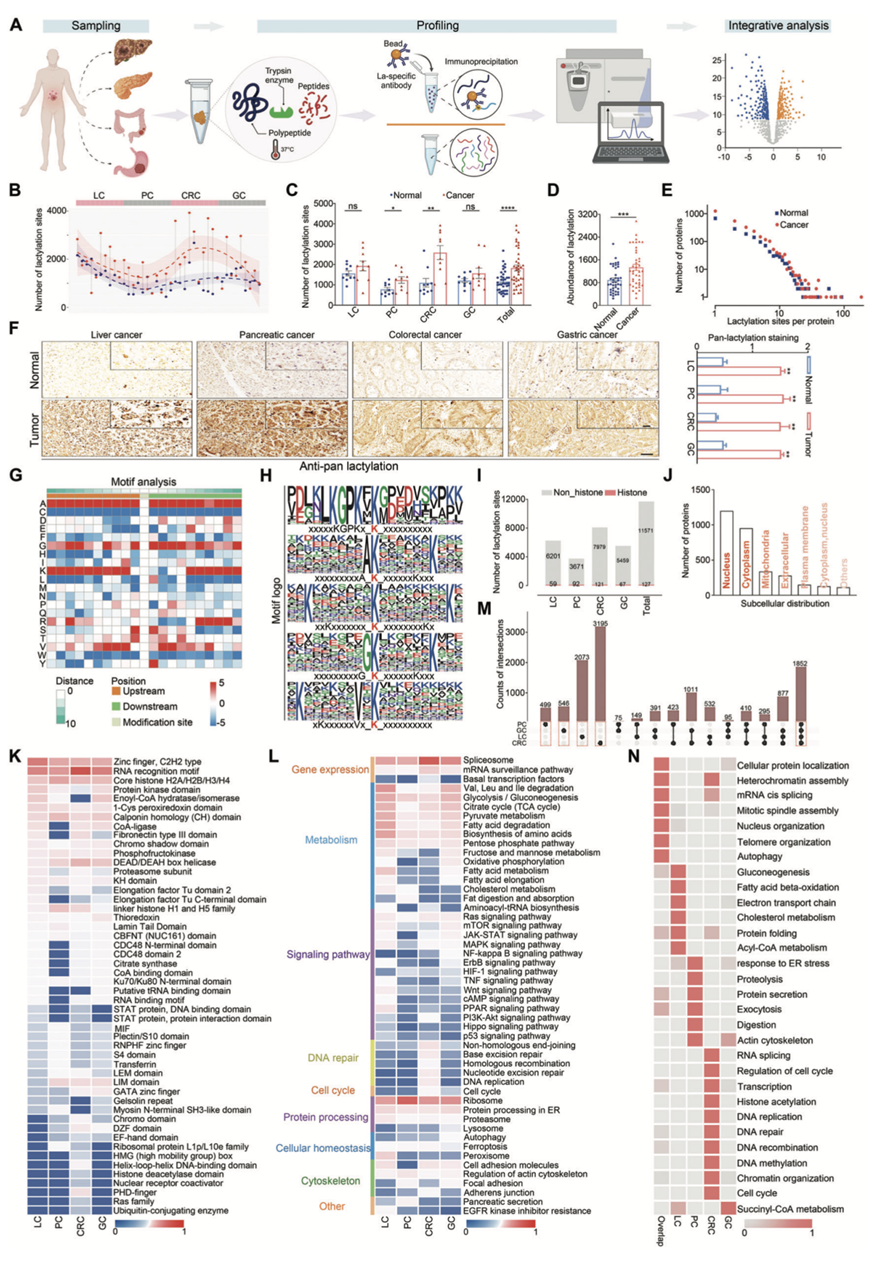
图1.胃肠道癌症蛋白质组分析。
(A) 实验示意图。(B) 胃肠道肿瘤和非肿瘤样本中 Kla 鉴定的概览。 (C) 胃肠道样本中 Kla 位点识别的箱线图。(D) 胃肠道样本中 Kla 总体丰度的定量分析。(E) 胃肠道肿瘤和 NAT 中每种蛋白质的 Kla 位点数量。(F) IHC 染色显示胃肠道癌症和 NAT 中的泛 Kla 水平。(G) Kla 位点 21 种氨基酸组成的热图,显示了乳酸化赖氨酸两侧特定位置中不同氨基酸的频率。(H)分析 Kla 上游和下游的十个氨基酸,并显示得分最高的前五个基序。(I) 在非组蛋白和组蛋白上检测到的 Kla 位点的数量。(J) 已鉴定的所有乳酸化蛋白的亚细胞定位。(K-L) 使用 DAVID 数据库对四个 GI 队列中乳酸化蛋白的富集进行蛋白质结构域和 KEGG 通路分析。(M) 从四个 GI 队列中鉴定出的所有 Kla 位点的交集。 (N) GO分析。
02
胃肠道癌症中 Kla 变异的综合乳酸组分析及其临床意义
乳酸组和蛋白质组数据集的重叠分析表明,两个数据集中有 53 种蛋白质显著失调(图 2A),他们将这些蛋白定义为胃肠道癌症相关蛋白,其中大多数的乳酸位点丰度变化大于相应的蛋白质丰度变化。细胞区室分析证实,53 种胃肠道癌症相关蛋白中有 20 种位于细胞质中,16 种位于细胞核中,15 种位于线粒体中,2 种位于细胞外,3 种位于血浆中(图 2B)。KEGG 通路分析表明,具有差异乳酸化位点的蛋白质分布在许多功能通路中,特别是代谢过程,包括脂肪酸代谢、糖酵解/糖异生和氨基酸代谢(图 2C)。除了几种代谢通路外,与剪接体和细胞周期/DNA 修复过程有关的差异表达蛋白(DEP)在胃肠道肿瘤中显著富集(图 2D)。亚型特异性通路富集分析清楚地展示了四种胃肠道癌症类型的分子特征,四种癌症代表性通路中乳酸化显著改变的蛋白质在标志性通路中发挥作用,包括剪接体、葡萄糖代谢、脂肪酸代谢、细胞周期和 DNA 修复(图 2E)。他们进一步根据肿瘤与 NAT 的比率和相应的蛋白质丰度将所有 Kla 位点分为四个象限,然后进行通路富集分析。由于肿瘤中乳酸水平较高,右两个象限(Kla 均上调)的 Kla 位点数量多于左象限(Kla 均下调)。在剪接体、代谢和细胞周期/DNA 修复通路中的大多数差异 Kla 位点都略有改变(图 2F)。这些结果进一步表明高乳酸环境在癌症标志性通路中的特殊调节作用。
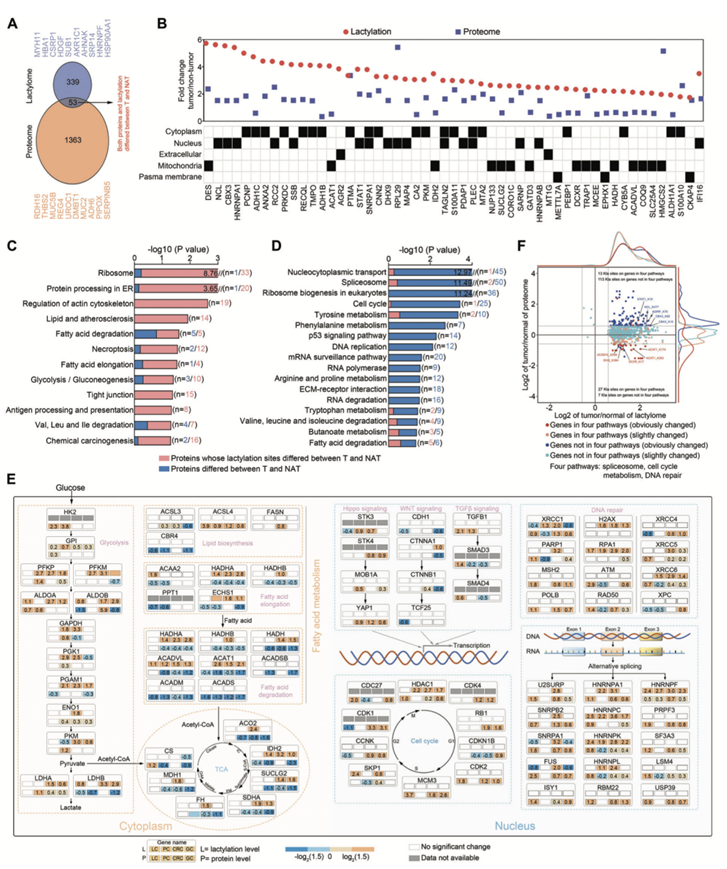
图2. 胃肠道癌症中 Kla 变异的综合乳酸组分析。
(A) 含有差异 Kla 位点的蛋白质(乳酸组)和 DEP (蛋白质组)的重叠。(B) 53 种胃肠道癌症相关蛋白的肿瘤和匹配 NAT 之间的差异倍数及其亚细胞定位。(C-D) 基于 DAVID 数据库进行通路富集分析。(E) 基于综合乳酸组分析的癌症标志性通路概述。(F) 象限图描绘了与 NAT 相比肿瘤中 Kla 位点的改变。
预后不良的胃肠道癌表现出侵袭性临床病理特征,例如肿瘤分期较高和肿瘤直径较大。因此,确定与这些侵袭性临床病理特征相关的 Kla 或蛋白质的特征具有临床意义。基因集富集分析确定了 31 条核心通路,其与代谢和基因调控相关的特征在 II 期和 III 期GI癌中富集,参与细胞粘附和细胞外基质组织的蛋白质在 I 期癌症中富集(图 3A)。他们确定了 103 个 Kla 位点和 762 种蛋白质可以区分组织病理学侵袭性肿瘤和侵袭性较低的肿瘤(图3B),进一步分析表明,401 个调节代谢重编程、剪接体和粘着斑的差异 Kla 位点在直径 > 5 cm 的较大肿瘤中富集(图 3C-D)。结合蛋白质组学数据分析显示,在较大的肿瘤中还发现了 252 个参与多种功能通路的 DEP(图 3E-F)。对这些差异Kla位点或DEP进行更严格、无监督的层次聚类,几乎完全将较大的肿瘤与直径≤5 cm的肿瘤区分开来(图3G)。此外,他们发现27个Kla位点和32种蛋白不仅在胃肠道肿瘤中存在差异表达,而且与肿瘤分期和直径均相关(图3H-I),这表明这些特征可能对胃肠道肿瘤的总体进展具有临床意义。
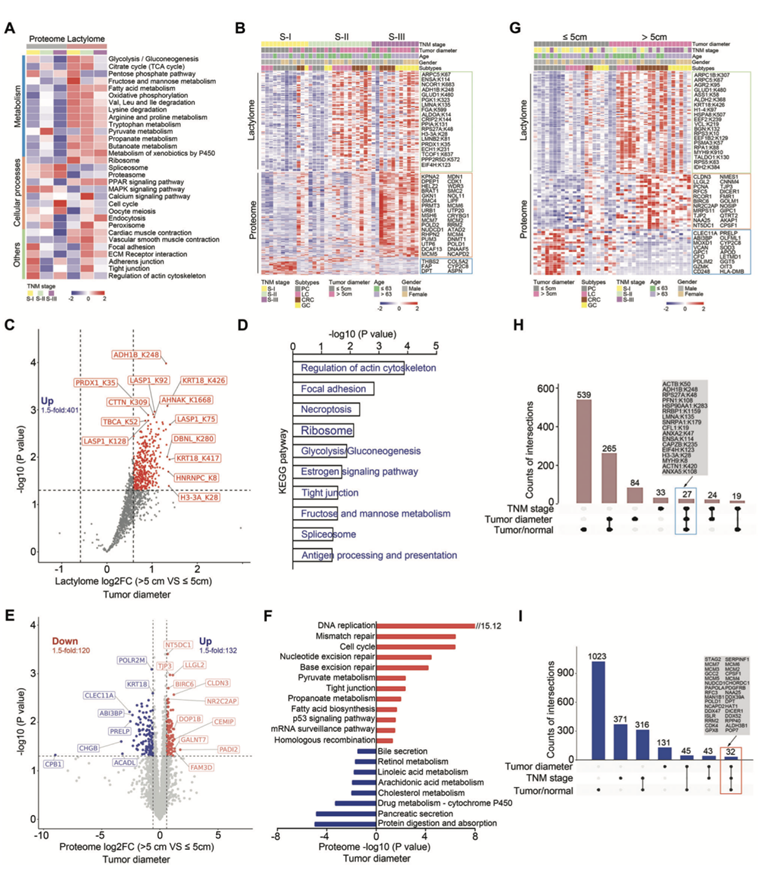
图3. 根据临床特征的乳酸组通路改变和差异表达。
(A) 不同肿瘤阶段改变通路的热图。(B) 与不同肿瘤阶段相关的 Kla 位点或蛋白质的层次聚类。(C) 火山图描绘了与肿瘤直径相比的差异 Kla 位点。(D) 条形图显示差异 Kla 位点的富集通路。(E) 火山图描绘了不同患者肿瘤的 DEP 。(F) 通路富集分析。(G) 差异 Kla 位点或 DEP 的层次聚类与所示临床病理特征的比较。(H-I) 与所示临床病理特征相关的差异 Kla 位点或 DEP的重叠。
03
组蛋白乳酸化的调控
已证实含有 Kla 的组蛋白对基因表达调控至关重要。在本项研究中,总共有 127 个 Kla 位点跨越不同的组蛋白(图 4A),并且在胃肠道肿瘤组蛋白中鉴定的 Kla 位点数量明显高于正常组织(图 4B),不同组蛋白的乳酸化水平在胃肠道癌中整体增加(图 4C)。对组蛋白中 127 个 Kla 位点进行无监督层次聚类将正常组织与胃肠道癌区分开来(图 4D),并且在胃肠道癌队列中发现了 15 个差异 Kla 位点(图 4E)。此外,15 个差异 Kla 位点中有 7 个位于组蛋白 H15 结构域,该结构域是染色质结构的重要组成部分(图 4F)。通过分析这些差异Kla位点的乳酸化水平与其他蛋白质组学数据的相关性,他们发现H1-0:K82或H1-4:K52乳酸化程度较高的肿瘤表现出多种功能通路的蛋白质组失调(图4G-H)。
他们还发现BRD2(一个溴结构域和末端外结构域家族成员)的表达水平与ELP3蛋白和组蛋白乳酸化丰度呈正相关,而SIRT3蛋白水平与大多数差异Kla位点呈负相关,表明这些酶可能调节组蛋白Kla水平,此外,BRD2在一定程度上可防止脱乙酰化(图4I)。
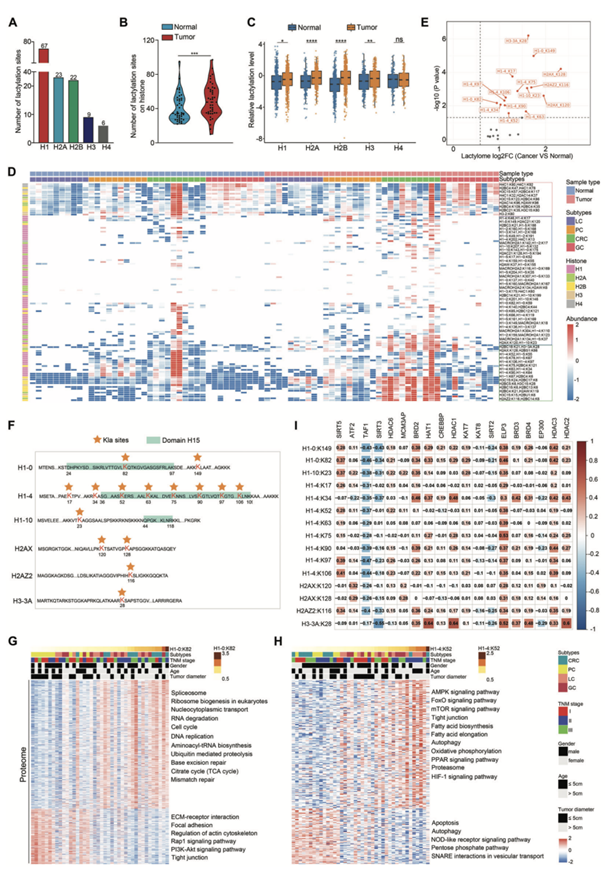
图4. 组蛋白乳酸化的调节。
(A) 胃肠道样本中不同组蛋白上检测到的 Kla 位点数量。(B) 直方图显示在胃肠道肿瘤或 NAT 中检测到的组蛋白 Kla 位点数量。(C) 显示了胃肠道肿瘤和 NAT 之间特定组蛋白的乳酸化水平变化。(D-E) 热图和火山图分析表明胃肠道癌症中不同组蛋白上的 Kla 位点发生了改变。(F) 组蛋白分子氨基酸序列中差异 Kla 位点的分析。(G-H) Kla 丰度与临床病理特征和蛋白质组分析的关联。(I) 组蛋白乙酰化酶和去乙酰化酶与组蛋白乳酸化水平之间的关联。
04
肿瘤 NAT 揭示的不同胃肠道肿瘤亚型特异性特征的比较
胃肠道癌症在细胞和组织学层面都是异质性疾病,为了阐明不同类型胃肠道癌症中蛋白质调控的泛癌症模式,他们使用模糊 c 均值算法根据蛋白质或 Kla 位点的改变进行聚类分析,并显示了六种模式。进一步对这些模式进行了GO分析和蛋白质结构域分析(图 5A-B)。基于乳酸组的聚类 1 突出显示了具有含有锌指结构域、14-3-3蛋白和硫氧还蛋白结构域的乳酸化蛋白。基于乳酸组的聚类 2、4 和 5,以及基于蛋白质组数据的聚类 1、4、5 和 6 显著富集了参与代谢的蛋白质,这些蛋白质与糖酵解、三羧酸 (TCA) 循环和脂肪酸代谢有关,表明这些聚类中的基因可能对调节胃肠道癌症代谢重编程至关重要。基于乳酸组的聚类6 和基于蛋白质组的聚类 3 高表达含有LIM结构域、锌指结构域和DEAD/DEAH框架解旋酶结构域的蛋白,这些蛋白参与 RNA 剪接和基因表达过程。这些结果进一步强调了乳酸组整合在识别肿瘤进展过程中分子稳态失调方面的重要性。
为了研究与特定肿瘤亚型相关的差异 Kla 位点,他们分析了在四种胃肠道癌症中发现的差异 Kla 位点的交集,发现 37 个 Kla 位点在所有四个队列中存在差异表达。重要的是,差异 Kla 位点在不同亚型中显示出更为离散的模式,其中仅在肝癌中发现 27 个位点,在胰腺癌中发现 37 个,在结直肠癌中发现 548 个,在胃癌中发现 69 个(图 5C)。GO分析表明不同类型的胃肠道肿瘤由蛋白质乳酸化背景下的不同分子机制驱动(图 5D)。他们还分析了在四种胃肠道癌症中发现的 DEP 的交集,在所有四个队列中发现了 46 个 DEP。此外,在肝癌中存在906差异表达基因(DEG),在胰腺癌中存在743 个DEG,在结直肠癌中存在835个DEG,在胃癌中存在542 个DEG(图 5E)。基于这些亚型特异性 DEP 的通路富集揭示了不同胃肠道癌症之间的特征(图 5F)。基于亚型特异性 Kla 位点和 DEP 的无监督层次聚类几乎完全将正常组织与胃肠道癌症分开,这表明这些特征可能与胃肠道癌症的肿瘤发生有关(图 5G-H)。
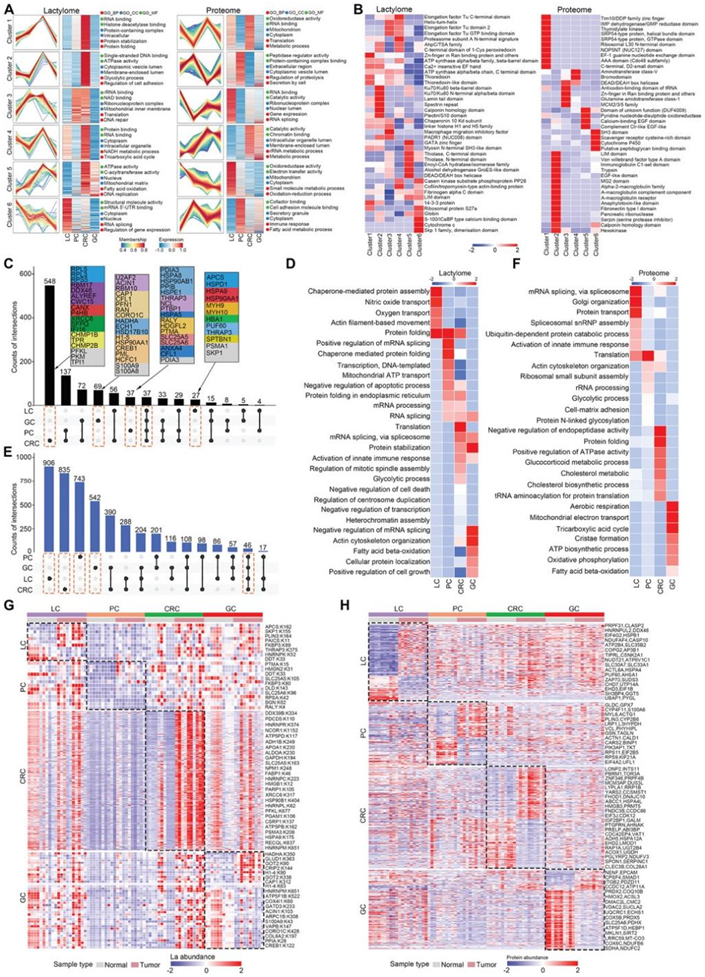
图5. 表征不同胃肠道肿瘤的亚型特异性特征。
(A) 不同胃肠道肿瘤中 Kla(左)或蛋白质(右)调控的泛癌模式。(B)乳酸化位点或蛋白质的蛋白质结构域富集分析。(C,E) 从四个胃肠道队列中鉴定出的差异 Kla 位点或 DEP的交集。(D,F)GO分析。(G-H) 每个胃肠道队列中亚型特异性 Kla 位点或蛋白质的层次聚类。
05
失调的 Kla 位点与胃肠道癌症中的基因调控密切相关
接下来,他们重点研究了四种胃肠道肿瘤中常见的差异 Kla 位点。无监督聚类显示,与正常组织相比,胃肠道癌症中 37 个 Kla 位点的水平持续上调(图 6A),并且这 37 个 Kla 位与调节 RNA 剪接、染色质结合、蛋白质结合和转录的多种分子功能和蛋白质复合物有关(图6B-D),表明赖氨酸乳酸化在基因调控中具有潜在的功能意义。此外,他们还分析了胃肠道肿瘤中 46 种常见的 DEP,通路分析显示这些蛋白质在许多必需的功能通路中富集。
由于基于胃肠道癌症中差异Kla位点的修饰组学强调了基因表达的失调,因此他们进一步分析了与RNA剪接、染色质结合或转录相关的12个Kla位点。胃肠道蛋白质组中广泛存在的Kla表明,这12个Kla位点在胃肠道肿瘤与NAT之间具有较高的频率和差异倍数(图 6E-F)。这12个Kla位点在不同物种间也具有保守性,部分位于蛋白质二级结构中(图6G)。BRD、HAT1 和 ELP3 蛋白水平与这 12 个 Kla 位点呈正相关,而 SIRT2 和 SIRT3 蛋白水平与大多数差异 Kla 位点呈负相关(图 6H-I)。总的来说,这些结果显示了不同胃肠道癌症之间 Kla 改变的一致性。
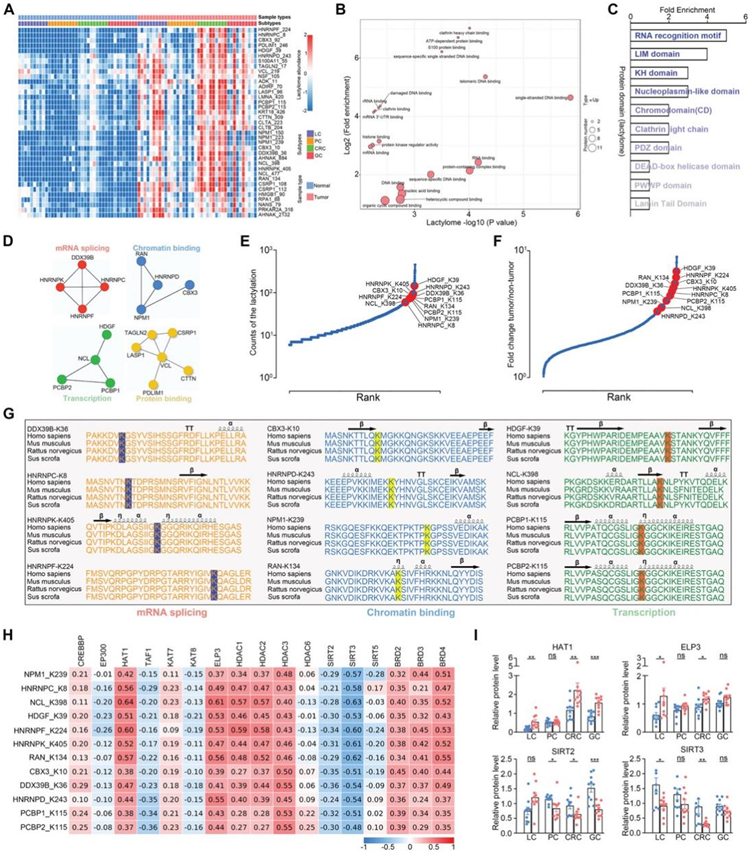
图6. 蛋白质赖氨酸乳酸化与胃肠道癌症的基因调控密切相关。
(A) 在所有四种胃肠道肿瘤中鉴定出的常见差异 Kla 位点的层次聚类。(B) 来自四个胃肠道队列的具有常见差异 Kla 位点。(C-D) 常见差异乳酸蛋白的蛋白质结构域和复合物富集分析。(E) Kla 位点相对于数据集中所有蛋白质的相对频率。(F) 胃肠道癌症中 Kla 水平相对于数据集中所有蛋白质的差异倍数。(G) 显示了来自不同物种的十二个 Kla 位点的保守性。(H) 乙酰化酶和脱乙酰化酶与所示乳酸化位点之间的关联。(I) 胃肠道肿瘤和 NAT中所示蛋白质的量化箱线图。
06
CBX3中的K10乳酸化促进CBX3与H3K9me3的相互作用和胃肠道肿瘤生长
染色体盒 3 (CBX3) K10的乳酸化修饰(CBX3 K10la)在胃肠道癌症中最为明显,该位点也位于 CBX3 的一个潜在小分子结合口袋中。为了确认胃肠道癌细胞中存在 CBX3 K10la,他们对瞬时表达的 CBX3 进行了免疫沉淀(图 7A-B),并通过 LC-MS/MS 分析了 CBX3 中的乳酸化修饰(图 7C-D)。此外,还制备了识别CBX3 K10la的特异性抗体,并用斑点印迹 (dot blot)和ELISA进行了验证(图 7E)。使用该特异性抗体的免疫印迹分析显示, CBX3在K10处乳酸化,进一步证实了CBX3 K10la的存在(图 7F)。此外,与正常上皮细胞相比,胰腺和结直肠癌细胞中的CBX3 K10la水平明显升高(图 7G-H)。
为了阐明可能影响细胞 CBX3 K10la 水平的因素,他们研究了参与乳酸生成和运输的基因的蛋白质组学数据。CBX3 K10la 水平较高的胃肠道肿瘤组织表现出己糖激酶 2 (HK2)、葡萄糖-6-磷酸异构酶 (GPI) 和丙酮酸激酶 M (PKM) 的蛋白质水平升高(图7I)。然而,在正常组织中并未观察到这种情况,可能是因为癌细胞主要依靠糖酵解来获取能量。
通过分析CBX3 和 CBX3 K10la 与其他分析数据的相关性(图 7J),他们发现具有较高 CBX3 K10la 水平的肿瘤表现出氧化磷酸化代谢相关蛋白质表达水平下调,以及剪接体、DNA复制和细胞周期过程的相关蛋白的上调。乳酸组分析表明,具有较高 CBX3 K10la 水平的肿瘤表现出核糖体、剪接体和代谢相关通路的上调。与 CBX3 及其 K10la 显著相关的蛋白质在负责基因调控的多个蛋白质结构域中富集(图 7K)。此外,CBX3 与 K10la 的分子对接分析表明 K10 乳酸化影响 CBX3 的构象(图 7L)。这些结果表明 K10 乳酸化在介导 CBX3 功能和调节胃肠道癌进展过程中的基因表达方面具有潜在作用。
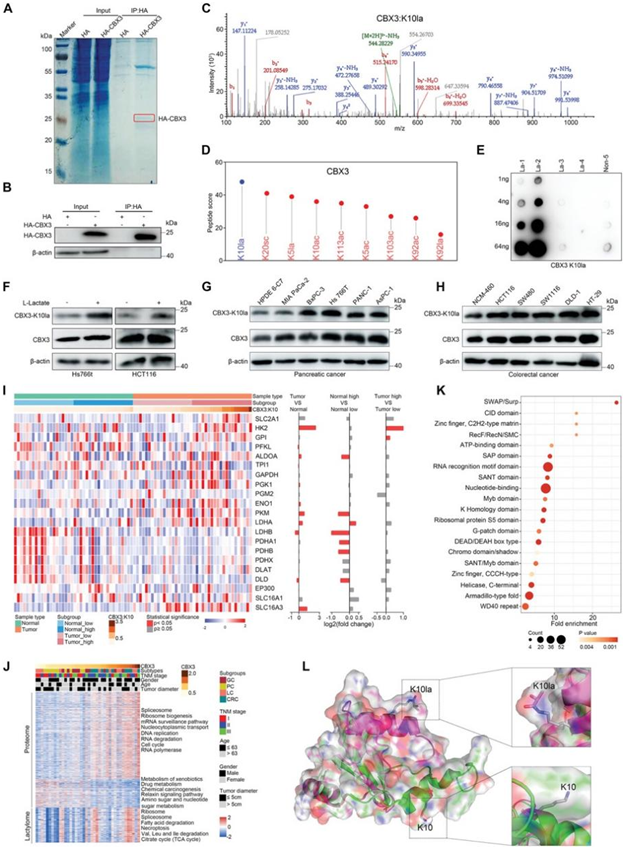
图7. CBX3 K10 乳酸化在胃肠道癌细胞中通常会升高。
(A-C) 通过免疫沉淀富集 Hs766T 细胞中与外源 HA-CBX3 的蛋白质相互作用,以进行 LC/MS-MS 分析。图中显示了含肽 K10 乳酸化的代表性光谱。 (D) 分析 CBX3 中具有不同酰化修饰的赖氨酸位点的丰度。(E) 生成兔多克隆抗体以特异性识别肽 NKTTLQ-K(Lactyl)-MGKKQN 中的K10la。 (F-H)免疫印迹分析。(I) 热图显示基于 CBX3 K10la 的涉及乳酸生成和运输的蛋白质的蛋白质组学表达。(J) CBX3 密度与临床病理特征和多组学分析的关联。(K) 基于与 CBX3 及其 K10la 水平相关的蛋白质的蛋白质结构域分析。(L) CBX3 结构与 K10 乳酸化或非乳酸化的分子对接分析。
CBX3 是一种高度保守的异染色质蛋白,通过结合 H3K9me3 特异性与染色质相互作用并调节基因沉默。免疫共沉淀分析进一步支持了这一结果,该分析显示在胃肠道癌细胞中内源性 CBX3 与 H3K9me3 结合(图 8A),并证明乳酸可以促进 CBX3 与 H3K9me3 的相互作用(图 8B)。此外,他们利用 CRISPR/Cas9 技术敲除胃肠道癌细胞中内源性 CBX3,然后用编码 WT CBX3 (CBX3 WT)或其突变体 K10R (CBX3K10R)的慢病毒表达质粒转染 CBX3 敲除 (CBX3KO)细胞,其中 Lys10 由精氨酸取代。免疫共沉淀证实了 H3K9me3 与 CBX3 WT结合,而与 CBX3 K10R结合的程度要小得多(图 8C),表明 K10 乳酸化促进了 CBX3 与 H3K9me3 的结合。
为了深入探究乳酸化如何促进 CBX3 与 H3K9me3 相互作用,他们利用分子动力学模拟比较了非乳酸化和 K10 乳酸化 CBX3 的结构动力学,并分析了 H3K9me3 肽的结合状态(图 8D)。分子对接分析表明,非乳酸化 CBX3 具有更动态的构象(从 300 纳秒开始收敛),而 K10 乳酸化从 200 纳秒开始保持稳定(图 8E)。此外,乳酸化 CBX3 中 H3K9me3 肽的均方根偏差 (RMSD) 在 7.5 Å-10 Å 之间波动,远低于非乳酸化 CBX3(12.5 Å-15Å)(图 8F)。乳酸化CBX3中H3K9me3肽段的旋转半径明显小于非乳酸化CBX3中H3K9me3肽段的旋转半径(图 8G),说明乳酸化CBX3结构模拟过程中波动幅度减小,这些结果说明CBX3在K10乳酸化过程中发生了有利于与H3K9me3肽段结合的构象变化。
为了探索 CBX3 K10la 与基因表达之间的相关性,他们对CBX3 K10R转染的胃肠道癌细胞进行了转录组分析(RNA-seq)。CBX3 KO和 CBX3 K10R细胞系中分别有 3251 个和 615 个DEG。上述两组之间的 DEG 交叉分析表明,CBX3 KO和CBX3 K10R细胞中均有 106 个基因失调(图 8H)。KEGG 通路分析表明,常见的 DEG 富集于许多标志性癌症通路中,包括糖酵解、细胞凋亡和对缺氧的反应(图 8I-J)。无监督层次聚类几乎完全将CBX3WT与CBX3KO和 CBX3K10R区分开来(图 8K)。这些结果表明 Kla 可以调节 CBX3 介导的转录过程,这对胃肠道癌进展至关重要。
CBX3KO 导致胃肠道肿瘤增殖明显停滞(图 8L),而 CBX3 K10R组的生长速度明显慢于 CBX3WT组(图 8M)。此外,将 CBX3KO或 CBX3 K10R转染的胃肠道癌细胞皮下注射到裸鼠体内。CBX3KO或CBX3 K10R表达阻碍了肿瘤生长(图 8N-O),这些结果表明 CBX3 上的乳酸化对胃肠道癌进展至关重要。
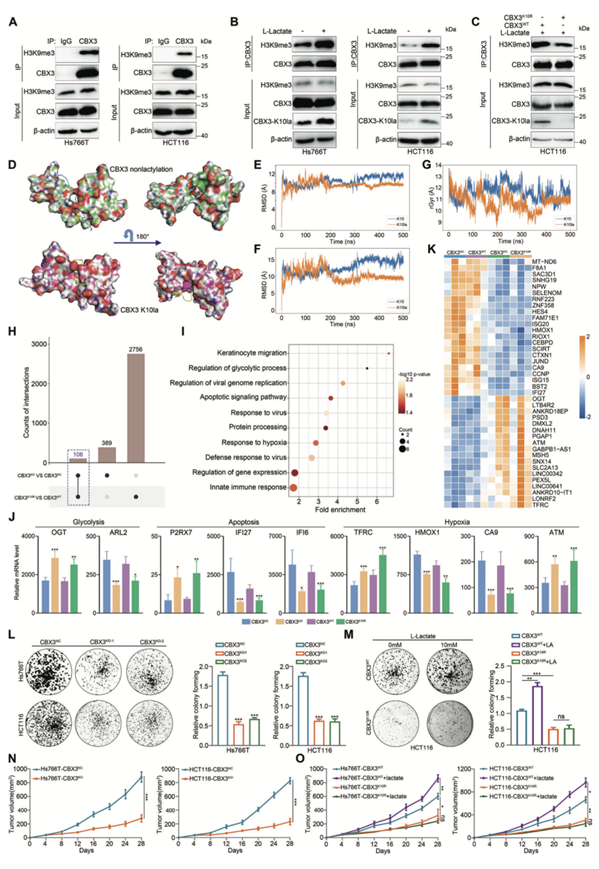
图8. K10 乳酸化促进 CBX3 与 H3K9me3 相互作用和胃肠道肿瘤生长。
(A-C)免疫共沉淀分析。(D) 基于 PDB 数据库中 CBX3-H3K9me3 复合构象对非乳酸化或 K10 乳酸化 CBX3 与 H3K9me3 肽进行分子对接分析 (PDB: 2L11)。(E) 非乳酸化和 K10 乳酸化 CBX3 的骨架均方根偏差 (RMSD) 随时间的变化。(F) H3K9me3 肽随时间的变化的 RMSD。(G) 非乳酸化或 K10 乳酸化 CBX3 中 H3K9me3 肽的回转半径 (rGyr) 分析。(H)DEG交集。(I) 通路富集分析。(J) CBX3 KO和 CBX3 K10R细胞中DEG及其相关的生物学通路。(K) CBX3 KO和 CBX3 K10R细胞中 DEP 的层次聚类。(L-M)克隆形成分析。(N-O) 肿瘤体积分析。
+ + + + + + + + + + +
结 论
本项研究对 40 名患有 GI 癌症的患者的癌组织和邻近组织进行了整体蛋白质组和乳酸化修饰分析,并确定了 11698 个 Kla 位点。乳酸化整合表明,Kla 影响涉及标志性癌症过程的蛋白质,包括表观遗传重组、代谢扰动和基因组不稳定性。此外,该研究揭示了 Kla 改变的泛癌症模式,其中 37 个 Kla 位点在所有四种 GI 癌症中持续上调并参与基因调控。进一步验证了 CBX3 在 K10 处的乳酸化修饰介导其与表观遗传标记 H3K9me3 的相互作用并促进 GI 癌症进展。
+ + + + +



 English
English


